Plant Count & Emergence for Microplot
1. Description
"Plant count and emergence for microplot" (Comptage de Plants et Émergence pour microparcelle) est un analytics qui détermine automatiquement le nombre de plantes et l'émergence pour les lignes de cultures dans les champs d'essai. Nous allons maintenant décrire son fonctionnement.
2. Fichiers d’Entrée
3. Workflow
Étape 1 - Dans un survey/relevé, choisissez l'analytics "Plant count and emergence fro microplot".
Étape 2 - Cliquez sur "LAUNCH".
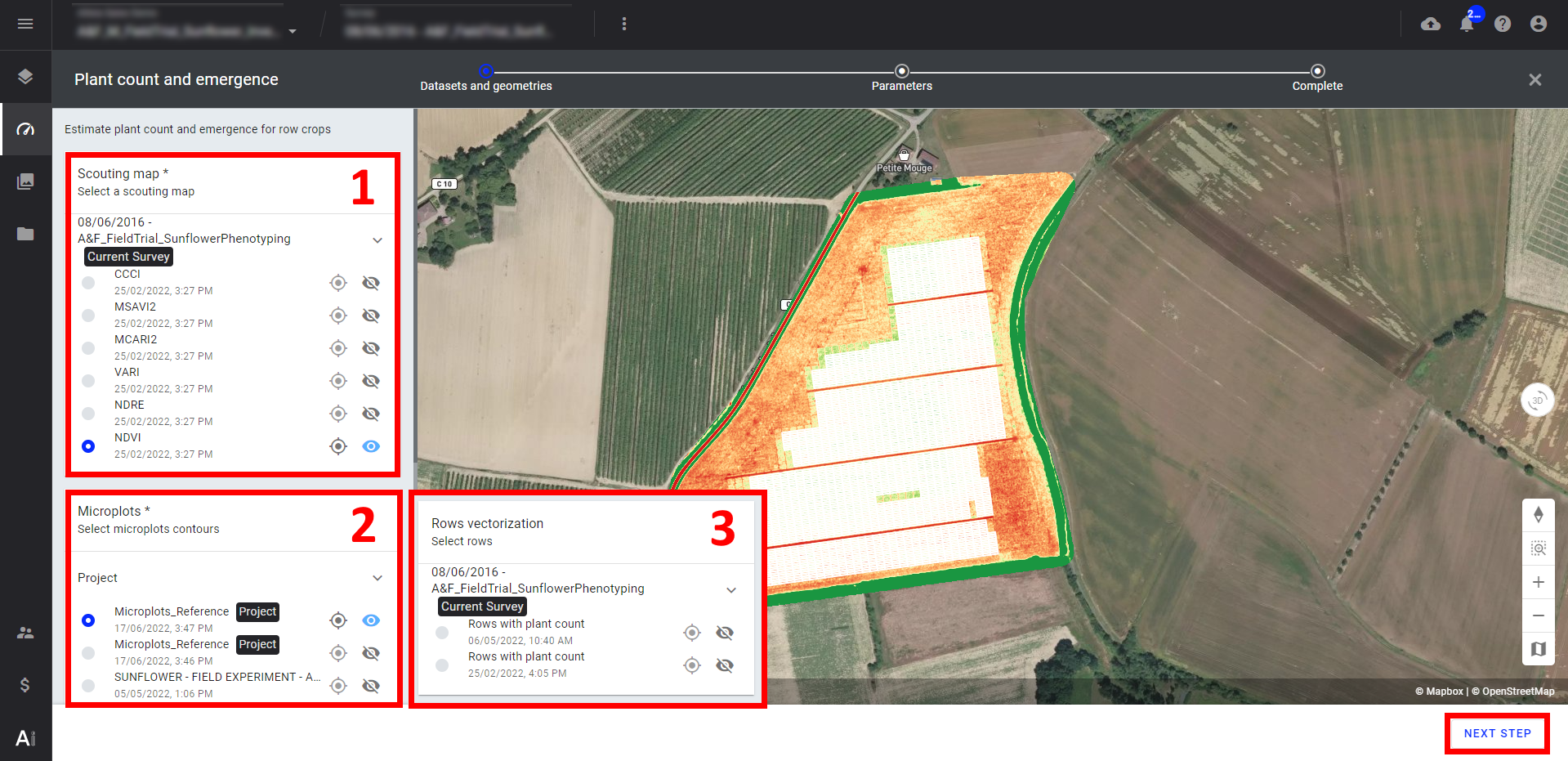
Étape 3 - Lorsque vous entrez dans la page analytique, sélectionnez le Spectral Index Map que vous utiliserez pour effectuer le comptage (1).
Étape 4 - Sélectionnez le vecteur Microparcelle qui sera utilisé (2).
Étape 5 - Sélectionnez les lignes vectorisées s'il y a lieu (3). Sinon, cette opération se fait automatiquement.
Étape 6 - Sélectionnez "NEXT STEP".
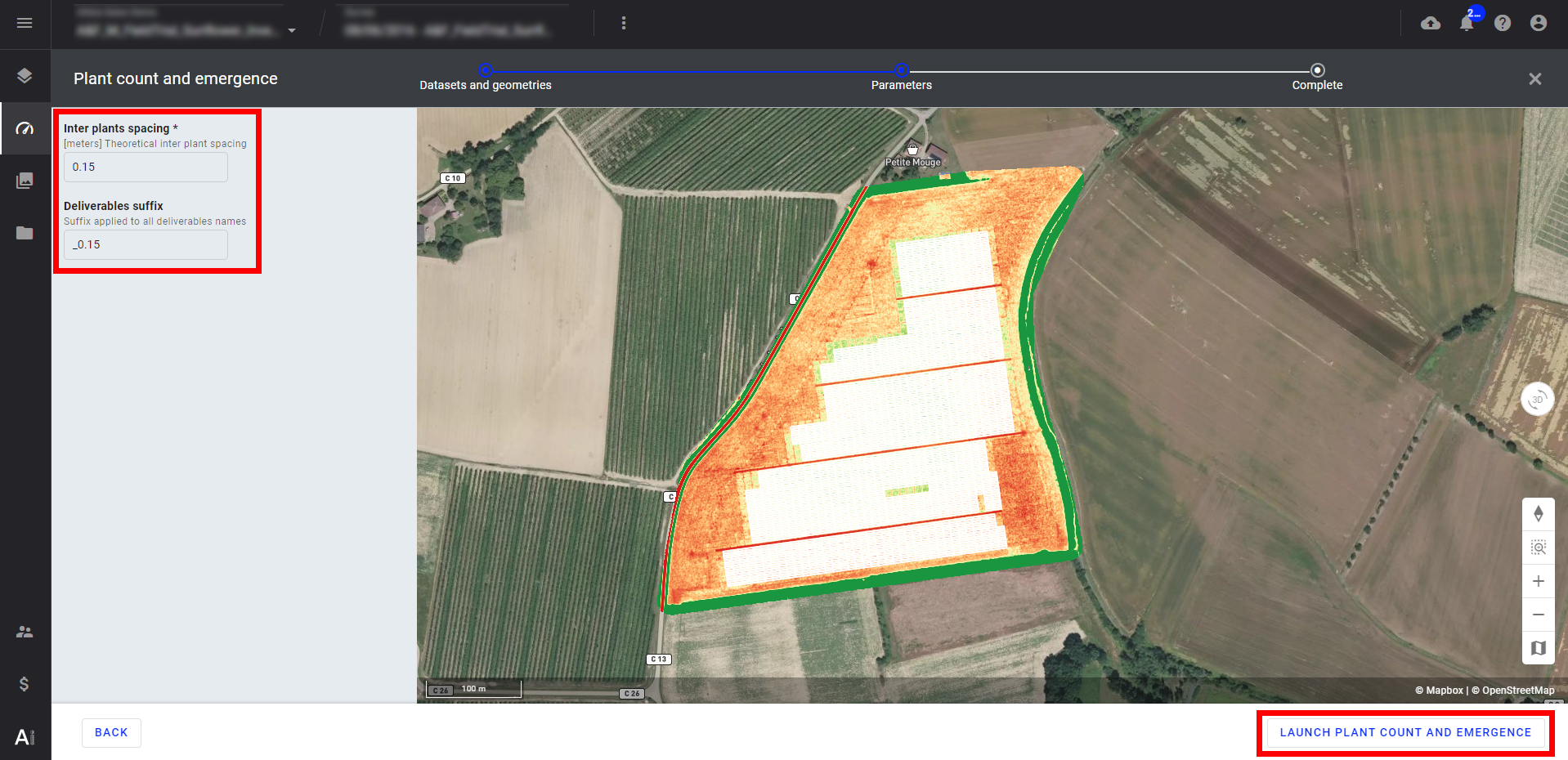
Étape 7 - Sur la page des paramètres, remplissez la section relative à l'espacement entre les plantes (inter-plants spacing), c'est-à-dire la distance entre deux plantes d'une même ligne.
Étape 8 - Saisissez un suffixe pour les produits livrables (facultatif). Par exemple : "_cultureA" produit "Plant count_cultureA.geojson", et cliquez sur "LAUNCH PLANT COUNT AND EMERGENCE FOR MICROPLOT".
 4. Résultats
4. Résultats
Dans le panneau des couches, affichez le dossier "Inventory" de la section "Survey Data", et affichez les couches "plant count " ou "gaps".
Si vous affichez la couche des écarts (Gaps), il est préférable d'afficher également la carte RVB pour identifier où se trouvent les écarts. Les écarts sont représentés par de petits points, et leur couleur dépend de la longueur de l'espacement mesuré. Reportez-vous à "Legend" pour les définitions des couleurs et des distances.
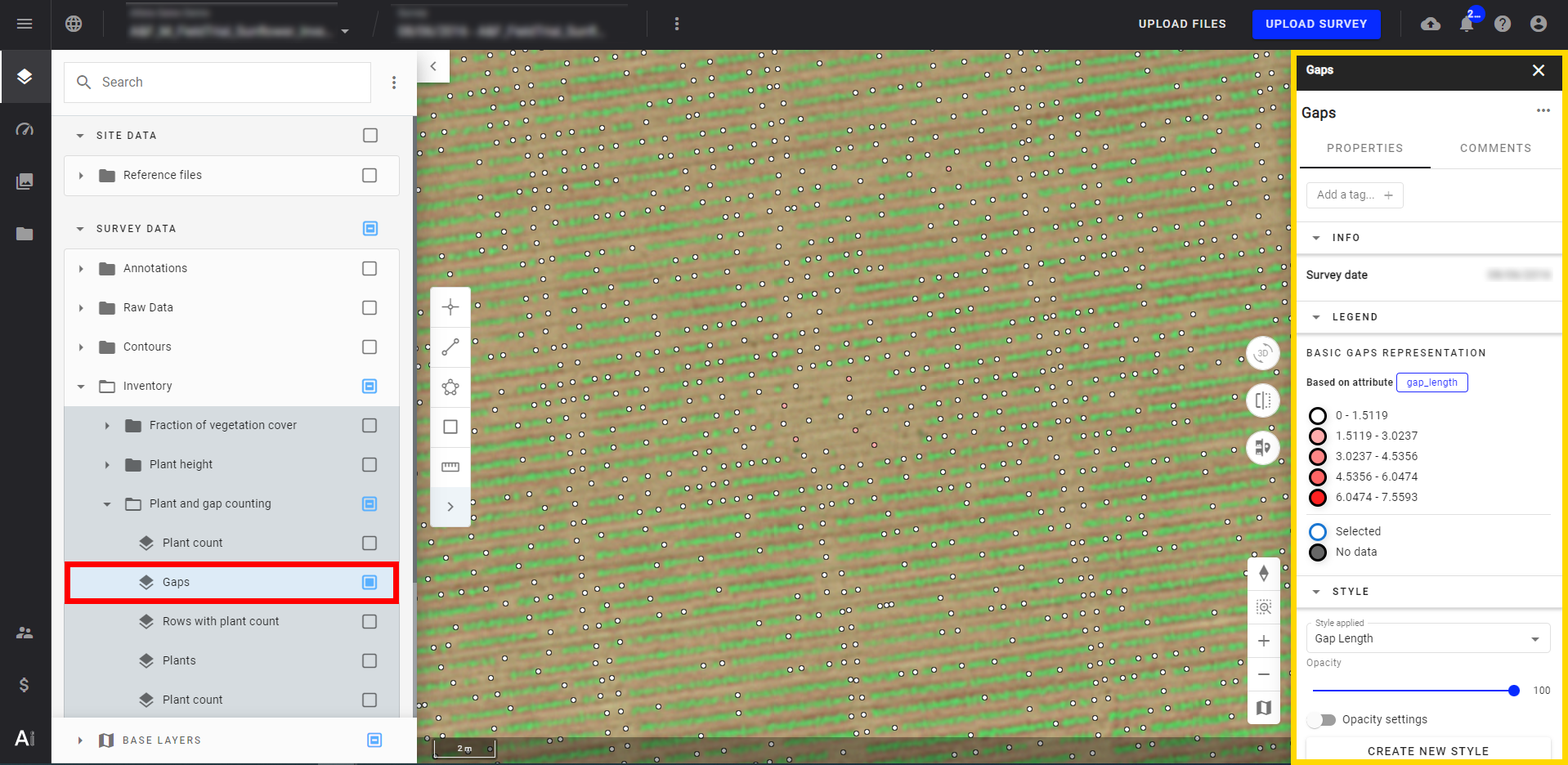
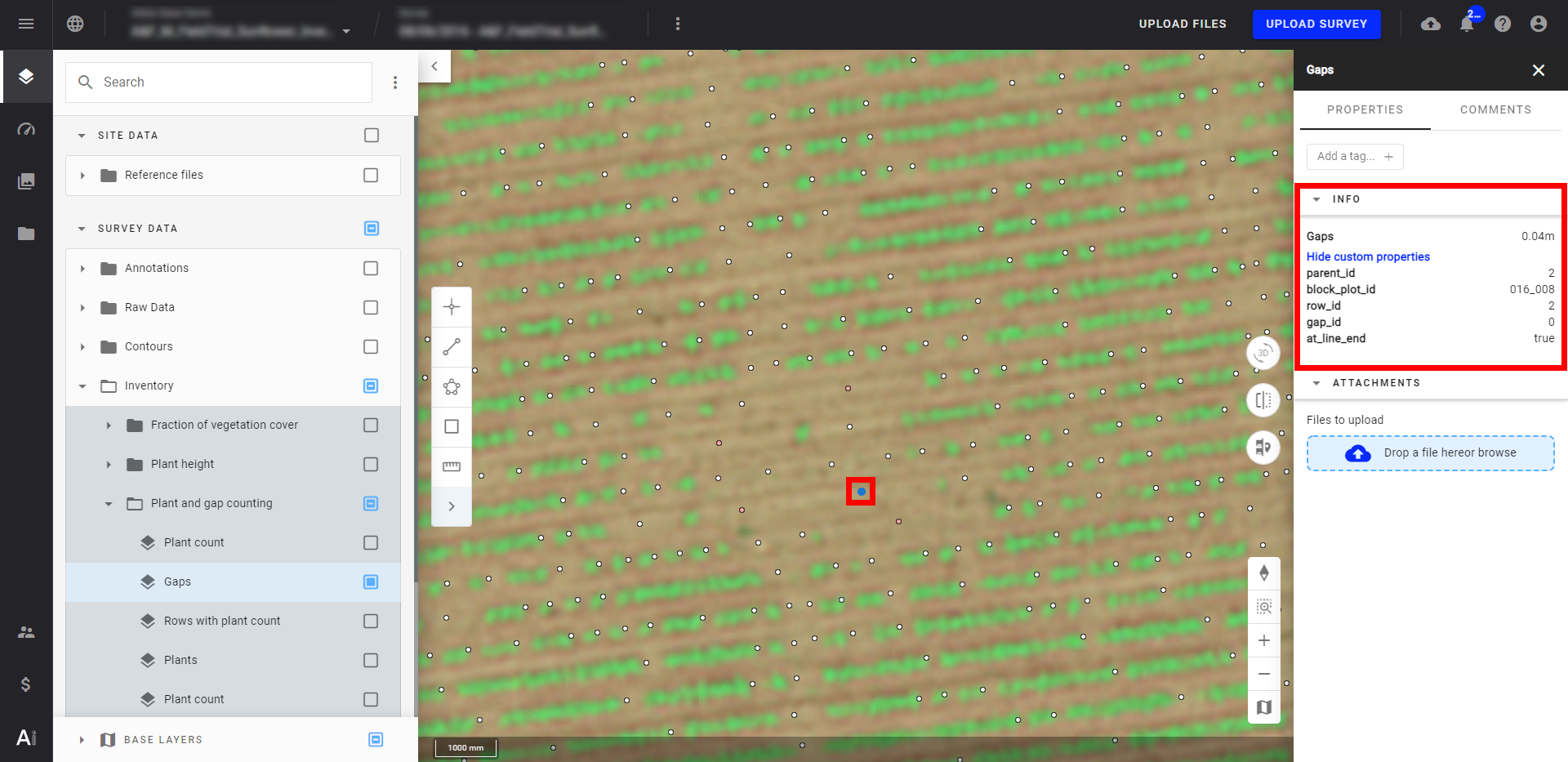
- De plus, en sélectionnant le point, la longueur de l'écart et d'autres attributs seront également affichés.
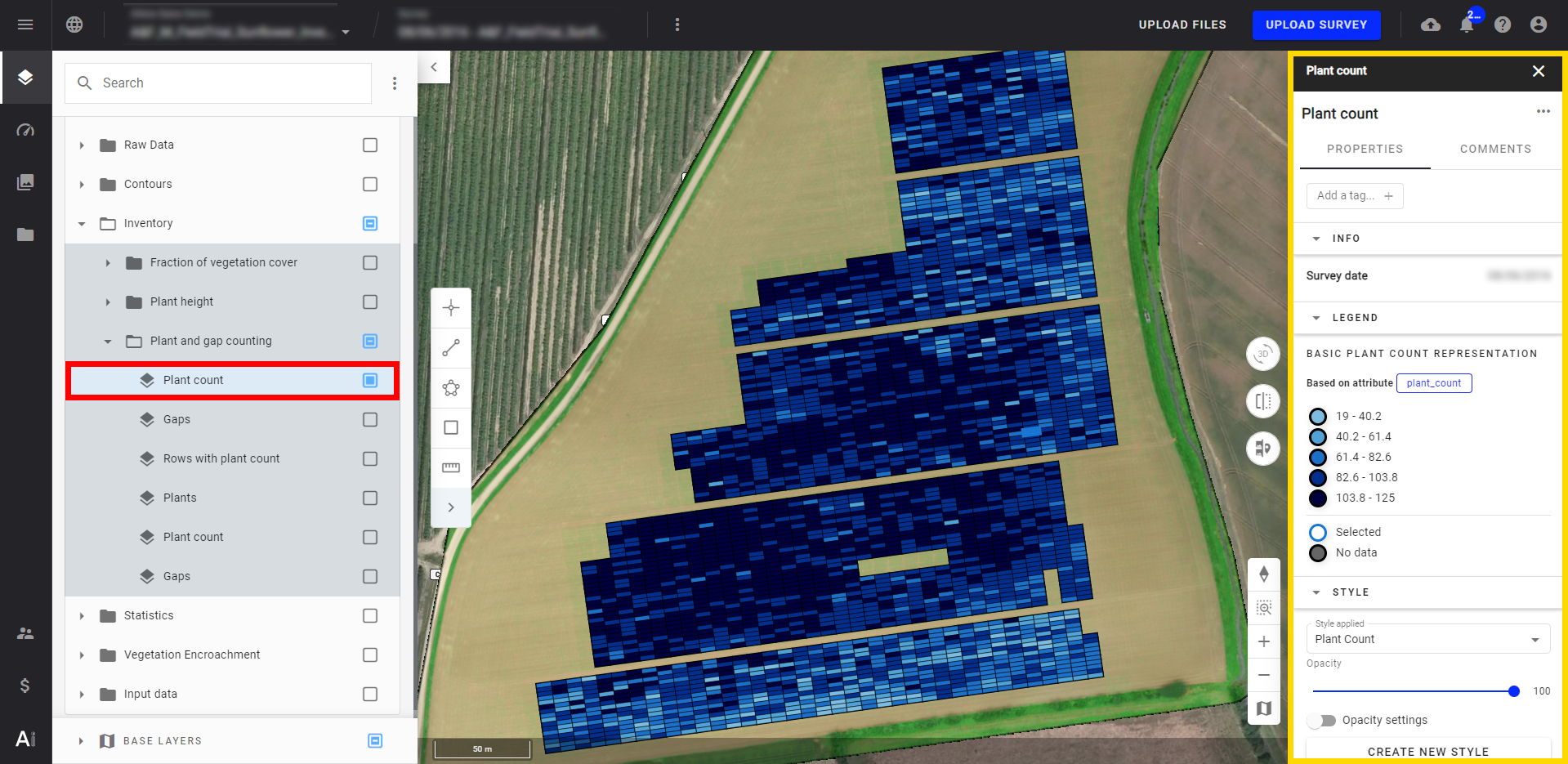
- Si vous affichez la couche de comptage des plantes (plant count layer), les microparcelles apparaissent. Les microparcelles seront également colorées en fonction de l'attribut sélectionné et de ses valeurs correspondantes.
- Cliquez sur une microparcelle pour afficher son mombre de plants et d'autres attributs.
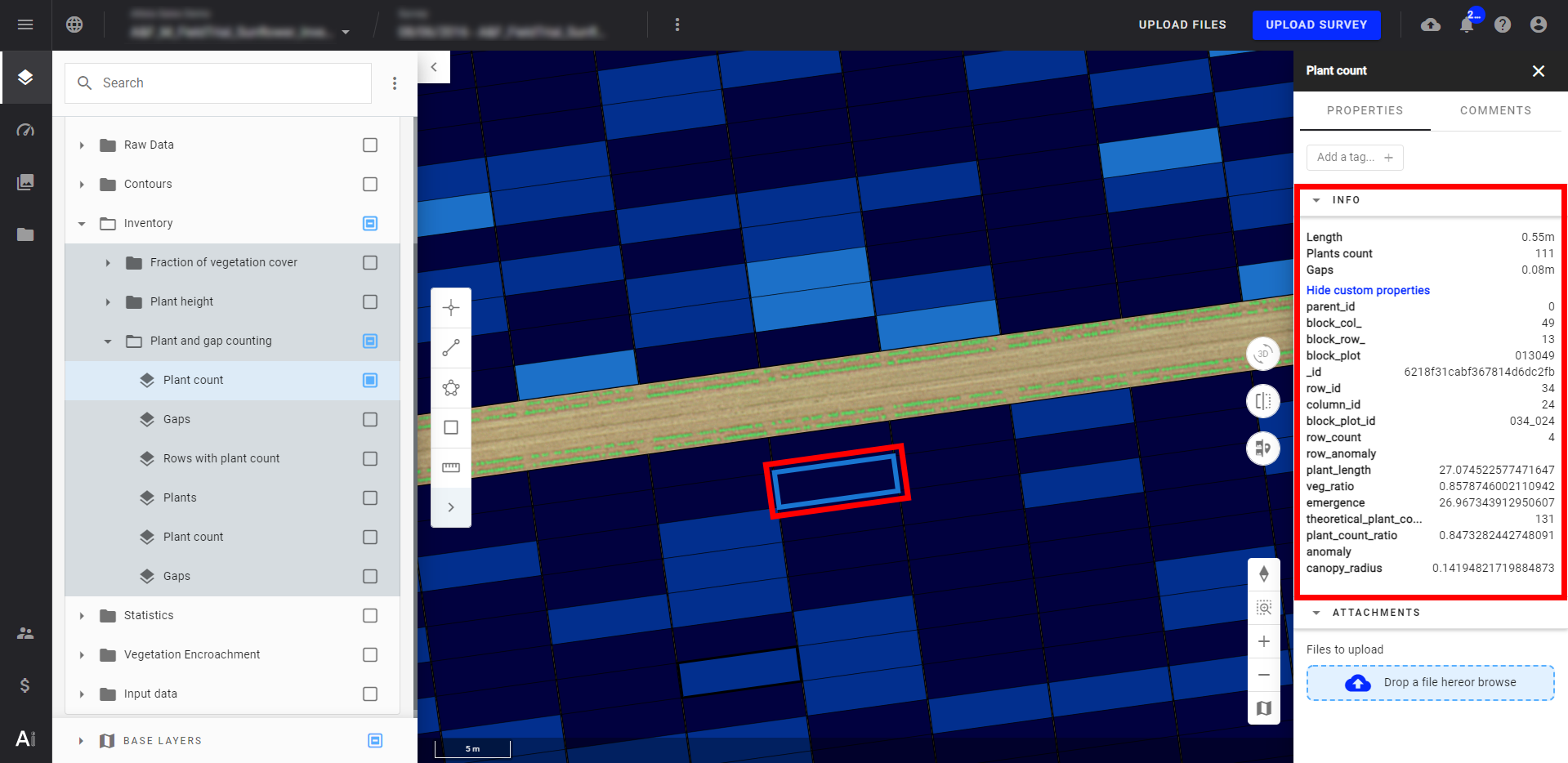
5. Produits livrables
Une fois l'analytics exécutée, les produits livrables peuvent être exportés dans deux formats standard, le format vectoriel tel que "geojson" et "csv".
Dans chaque format, vous pouvez trouver les attributs suivants:
6. Vérifier la qualité des résultats
Cette section permet d'identifier de potentielles anomalies sur les parcelles d'essais.
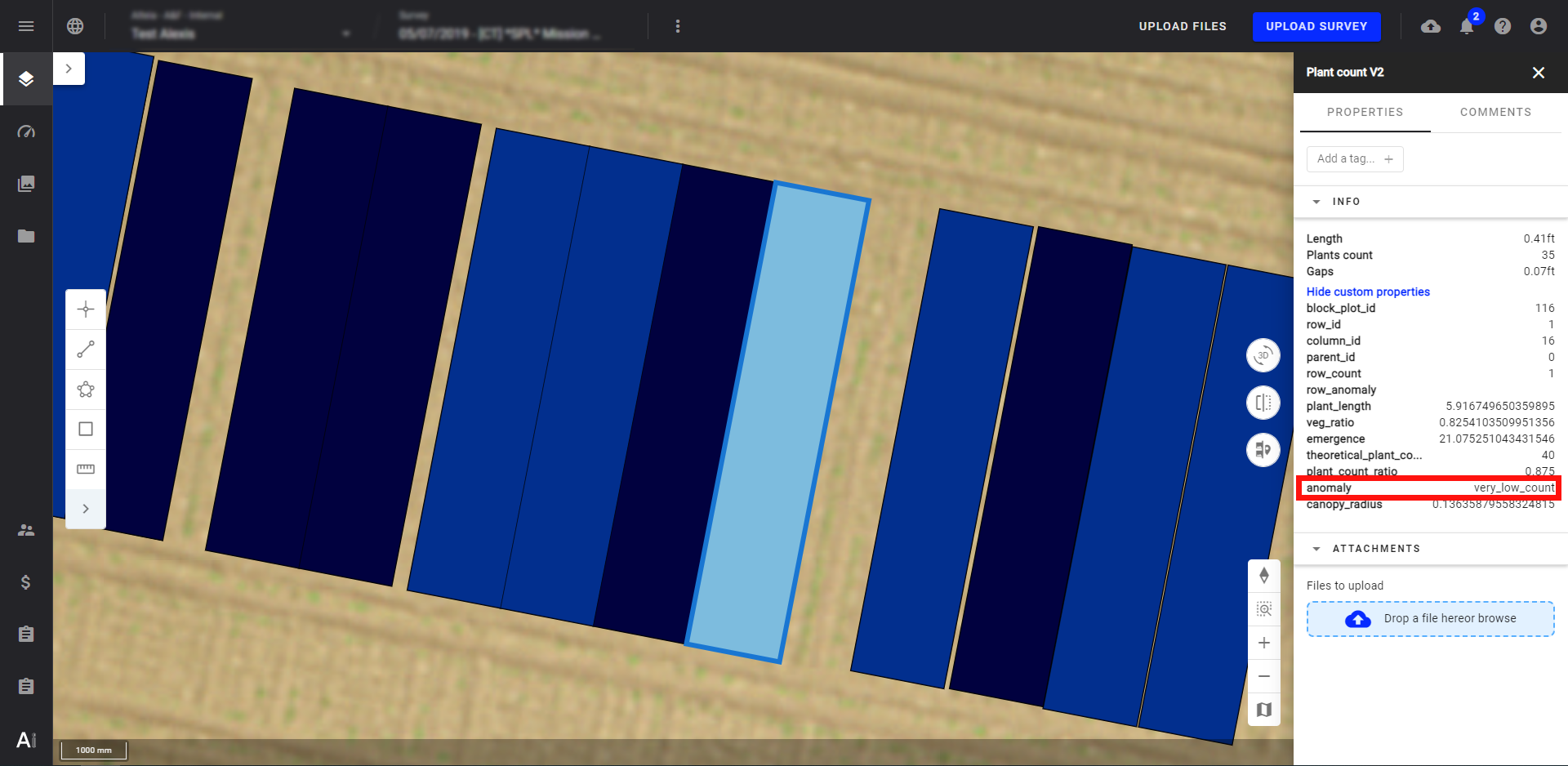
6.1 Vérifier l'attribut "anomaly"
Dans le vecteur de comptage de plants livré par Alteia, cliquez sur une microparcelle pour identifier si une anomalie est détectée. Vous pouvez également extraire le fichier CSV pour obtenir un rapport des anomalies relatif à toutes les parcelles.
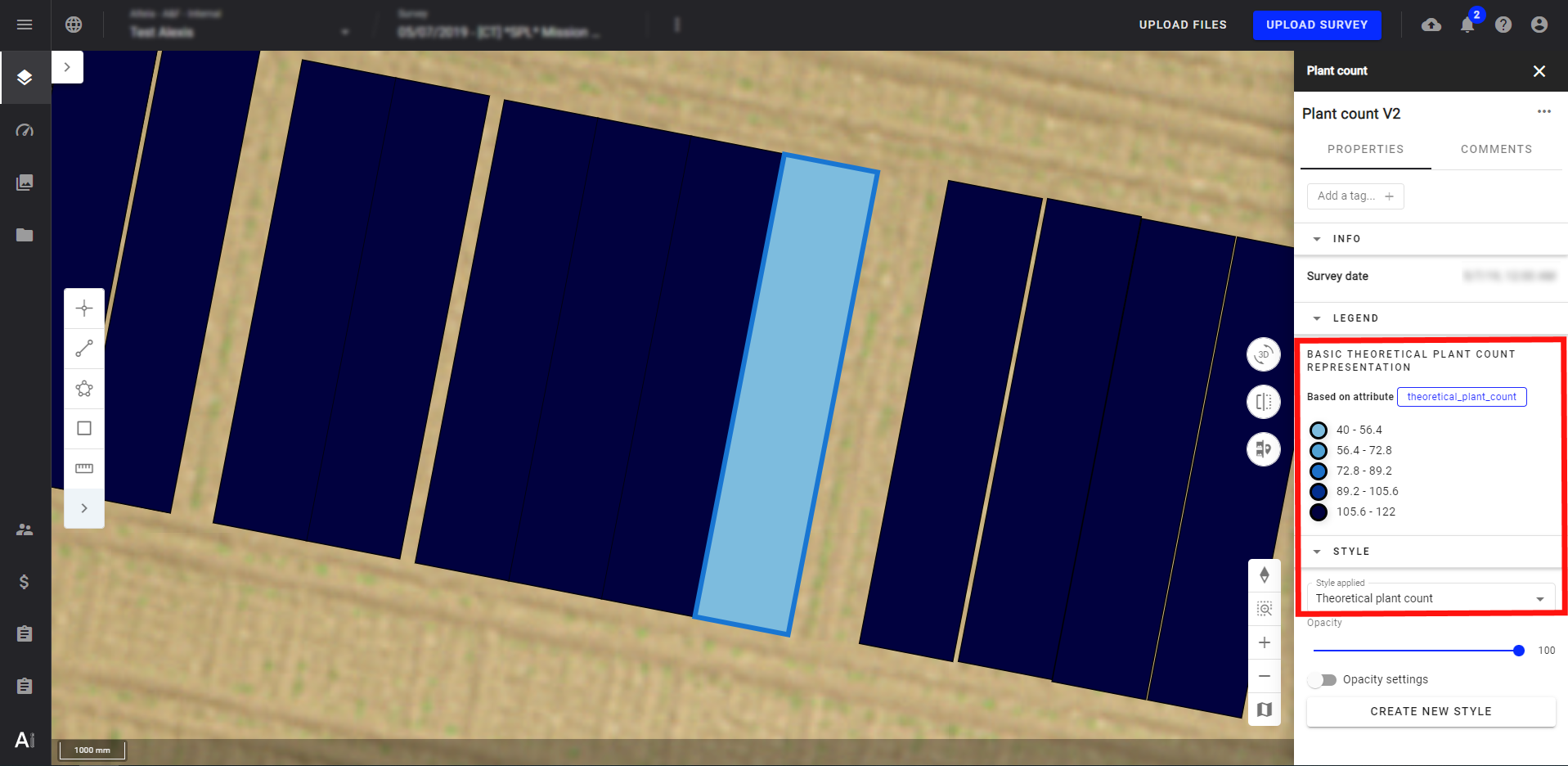
6.2 Verifier l'attribut "theorical_plant_count"
Si les valeurs sont très différentes entre le vecteur de comptage de plants livré et votre comptage théorique des plants, il existe probablement une anomalie.
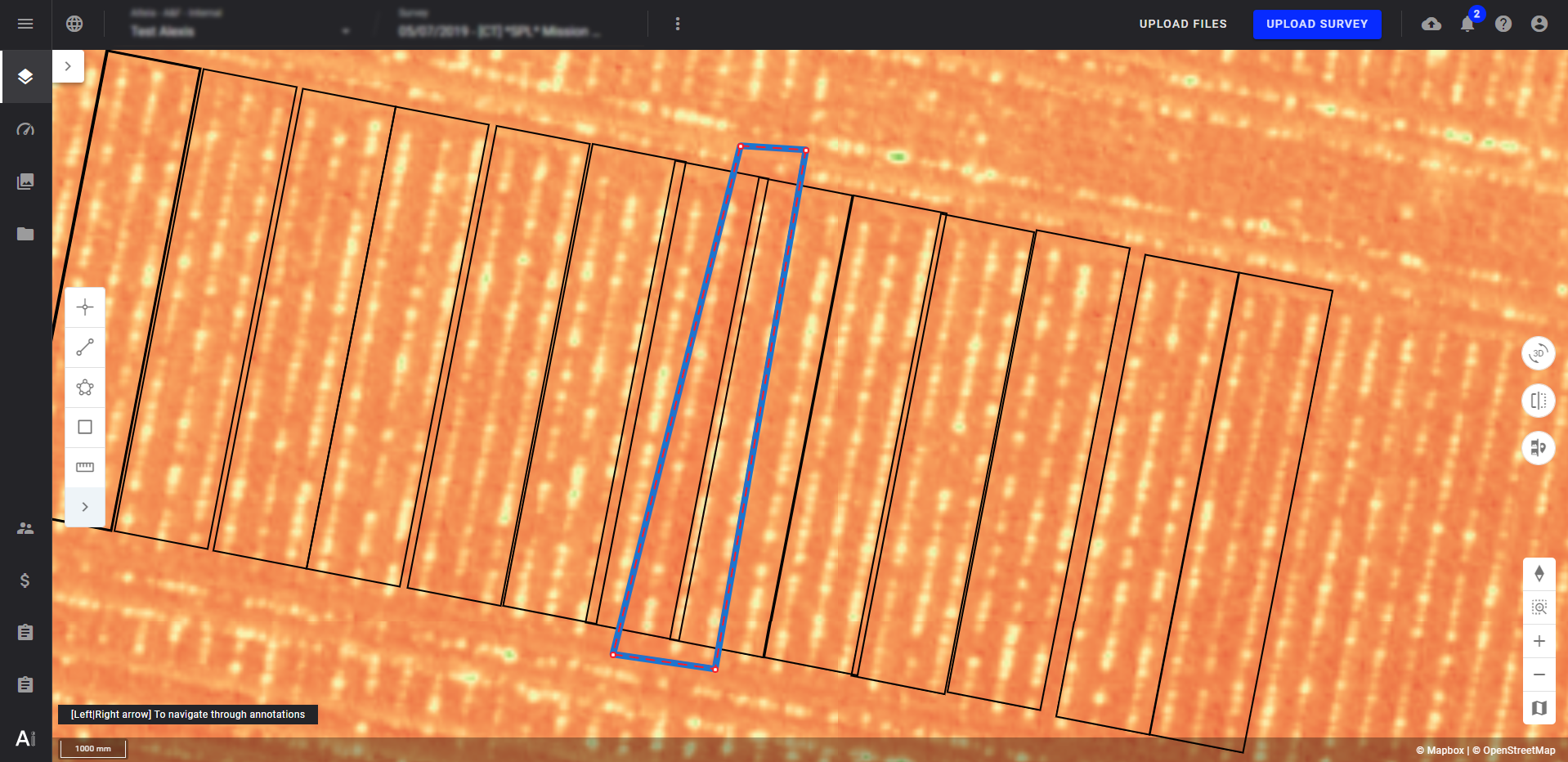
6.3 Vérifier le positionnement des vecteurs de microparcelles
Si le contour de la parcelle n'est pas bien situé autour des plants, les résultats peuvent être affectés. Utilisez une carte de repérage (comme le NDVI) en arrière-plan afin de mieux identifier les rangées de plants et d'effectuer des actions correctives.
L'exemple ci-dessous montre une superposition de microplots au niveau d'une rangée de plants. Les plants présents sur ce rang sont donc difficilement comptabilisés, ce qui affecte les résultats livrés.